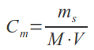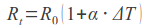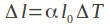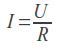W 2012 roku zespół M. W. Coverta opublikował kompletny model funkcjonowania komórki M. Genitalium. Po raz pierwszy możliwe stało się symulowanie działania genów oraz procesów bichemicznych zachodzących podczas reprodukcji organizmu. Jak do tego doszło?
W 1984 roku Harold Morovitz (wówczas z Yale University) rozpoczął badania nad stworzeniem modelu bakterii z rodzaju mycoplazma. Do badań wybrał M. genitalium – najprostrzą bakterię, z którą biolodzy pracowali już od dłuższego czasu. Pierwszym etapem badań było zsyntetyzowanie jej genomu – dokonał tego w roku 1995 J.Craig Venter i jego współpracownicy z The Institute for Genome Research (TIGR). Okazało się, że posiada ona 525 genów, z czego 400 ma kluczowe znaczenie dla podtrzymania życia (dla porównania ludzka komórka zawiera przeszło 20 000 genów). Rozpoczęto badania. Pierwsze symulacje prowadzono w całych koloniach komórek.
W 2008 roku M. W Covert wpadł na pomysł skonstruowania modelu pojedynczej komórki. Dokonał analizy wszystkich informacji zgromadzonych dotychczas w pracach naukowych teoretycznych, badaniach genetycznych i wcześniejszych doświadczeniach w modelowaniu życia M.genitalium. Mimo iż bakteria ta jest stosunkowo prostym organizmem, odtworzenie jest funkcjonowania okazało się poważnym wyzwaniem. Naukowcy opracowali ponad 1900 wirtualnych parametrów. Każdy z procesów biologicznych ujęli jako 28 osobnych „modułów“ zarządzanych przez własne algorytmy. Podczas wykonywania programu moduły w przypadkowej kolejności symulowały jednosekundowy etap życia bakterii. Po zakończeniu symulacji pierwszego jednosekundowego etapu, program, uwzględniając wyniki przeprowadzonych obliczeń, aktualizował dane. Stanowiły one punkt wyjścia do symulacji drugiego jednosekundowego etapu. Taki cykl był powtarzany do momentu podziału komórki, jej obumarcia lub do chwili, gdy komórka osiągała nierealistyczny wiek. Badania nad modelem trwały około cztery lata. W tym czasie zespół doskonalił model usuwając pojawiające się błędy programowe.

Źródło: Lee R, Karr JR, Covert MW. „WholeCellViz: data visualization for whole-cell models"BMC Bioinformatics. 2013. 14: 253.
red. Maja Haczyk, Biotechnologia.pl
Dalsza część artykułu dostępna TUTAJ